Organisation des données
L'un des principaux intérêts de l'utilisation de système de gestion et stockage de données réside dans la formalisation de ces données selon des standards communs établis. Cette page vous présente le format de classification utilisé par OMERO et donc par notre solution ainsi qu'un standard concernant les métadonnées associées à vos données de microscopie.
Organisation des répertoires et fichiers
Afin de respecter les conventions internationales en matière d'accessibilité et de réutilisation des données scientifiques (voir principes FAIR), une arborescence standardisée est imposée: la structure ISA (Investigation/Study/Assay). Elle fonctionne de cette façon-là:
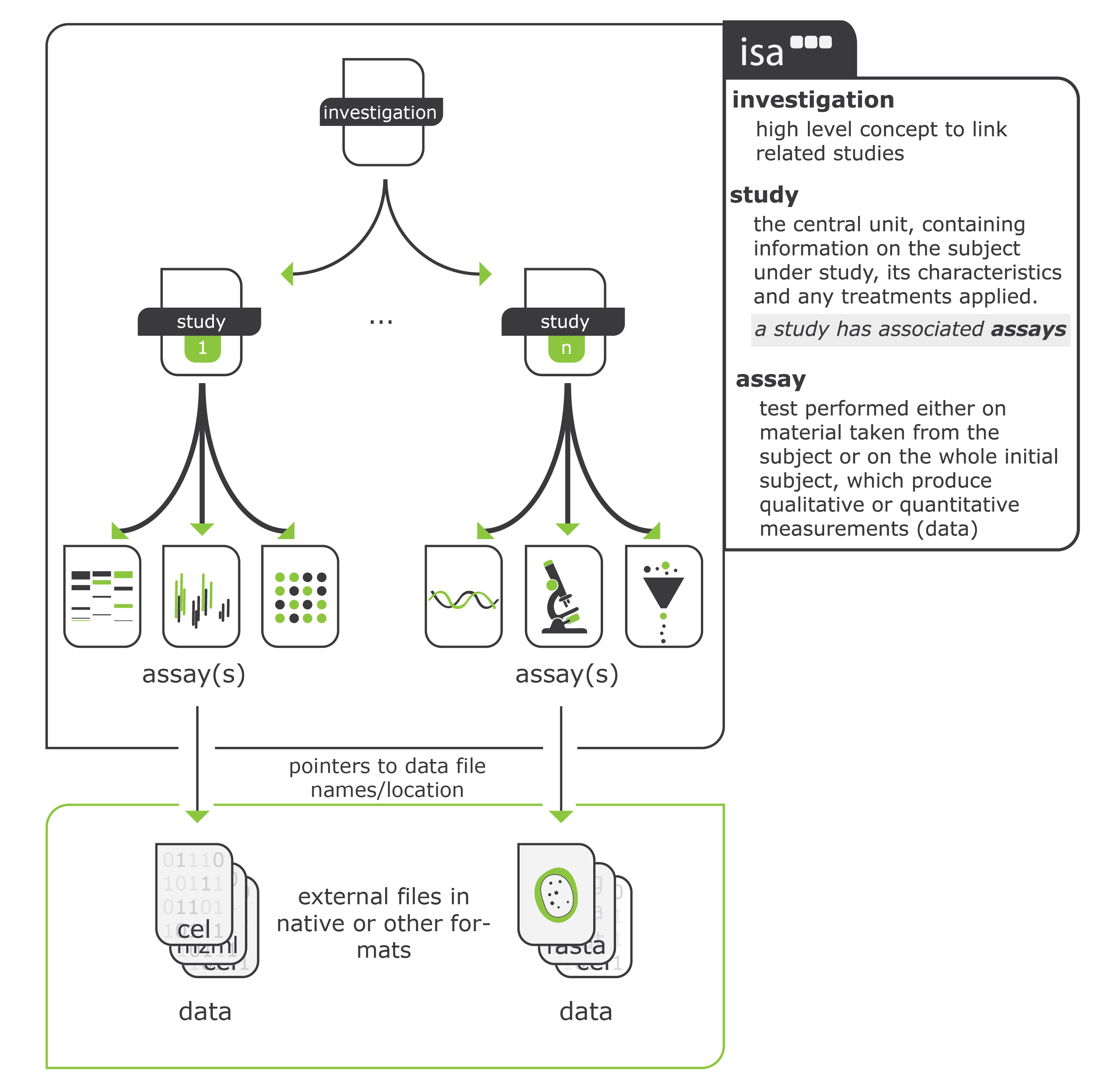
Dans OMERO, un Groupe correspondra à une investigation (un projet de recherche), un Projet correspondra à une étude (Study), un Dataset correspondra à un dosage (Assay).
Déposer les images dans une hiérarchie de répertoires "Nom de l'Investigation/Nom de l'Etude/Nom du Dataset" sur le répertoire tampon (Nom à définir) dans un répertoire nommé avec votre identifiant OMERO.
Le nom du projet de recherche ("investigation") doit évoquer la question de base, les principaux objectifs du projet de recherche, mais être suffisamment concis pour être utilisé comme nom. Il sera préférable d'utiliser un acronyme ou un numéro de projet, à la discrétion de la nomenclature utilisée par l'équipe de recherche en question. Mentionner s'il s'agit d'un projet dans le nom. Au pire, utiliser une composition de 2 ou 3 mots-clés cernant bien le thème du projet.
- Exemples de questions scientifiques de base:
- Rôle de la protéine X dans l'apparition du cancer Y
- Est-ce que la voie métabolique Z a un rôle dans l'apparition du cancer Y
- Exemple de nomenclature:
- "Radiothérapie interne pour le traitement de la leucémie aigüe myéloblastique" -> Projet_24P02 -> Dans ce cas, utiliser le numéro de projet + éventuellement un acronyme (RITLAM?)
- ex: Projet_24P02_RITLAM (acronyme)
- ex: Projet_LAM_radiotherapie (système de mots-clés)
- ex: Projet_24P02_LAM_radiotherapie (systèmes de mots-clés + numéro)
- "Radiothérapie interne pour le traitement de la leucémie aigüe myéloblastique" -> Projet_24P02 -> Dans ce cas, utiliser le numéro de projet + éventuellement un acronyme (RITLAM?)
Rappel
Ne pas utiliser de caractères spéciaux (ponctuation, espace, accents). Utiliser le _ (underscore) pour remplacer les espaces comme dans les exemples.
Le nom d'une étude ("Study") doit évoquer une sous-question, une hypothèse de départ dans la résolution de la question de base, mais être suffisamment concis pour être utilisé comme nom.
Il sera préférable d'utiliser un acronyme ou un numéro de projet, à la discrétion de la nomenclature utilisée par l'équipe de recherche en question. Mentionner s'il s'agit d'une étude dans le nom. Au pire, utiliser une composition de 2 ou 3 mots-clés cernant bien le thème de l'étude (en évitant de faire doublon avec les mots-clés du projet). - Exemple de sous-question de projet: - Est-ce que la protéine X à un rôle inducteur/inhibiteur/cofacteur dans le pipeline Z impliqué dans l'apparition du cancer Y?
Les expériences ("Assays") contenus dans une étude correspondent à des expériences individuelles (observations microscopiques, profils de transcription en réponse à l'ajout ou une modification d'un élément) destinées à résoudre la sous-question.
Pour le nommage, il sera préférable d'utiliser une description rapide du caractère observé et de la modification, à la discrétion de la nomenclature utilisée par l'équipe de recherche en question. Mentionner s'il s'agit d'un "assay" dans le nom. Au pire, utiliser une composition de 2 ou 3 mots-clés cernant bien le paramètre étudié pour l'expérience (en évitant de faire doublon avec les mots-clés du projet (investigation) et de l'étude (study)).
La composition exacte des noms permet une certaine liberté d'imagination et d'abstraction de la part de l'utilisateur, mais doit dans la mesure du possible respecter ces contraintes. Ce lien est une bonne piste de départ.
Note
Le serveur n'est pas un dépôt d'images pour téléchargement: il est demandé de ne pas dupliquer les images à destination d'une tierce personne dans le serveur (par exemple pas de répertoire "pour_machin"). La consultation d'images et l'interaction avec les autres utilisateurs se feront via OMERO et SFTP-Go.
Métadonnées à récupérer (dans la mesure du possible) - le standard REMBI
Définition
Quand on produit des données biologiques — images de microscopie, mesures, séquences, résultats expérimentaux — on pense d’abord aux données elles-mêmes. Mais sans leur contexte, ces données perdent rapidement leur sens. C’est précisément ce rôle que jouent les métadonnées. Les métadonnées sont tout simplement les informations qui décrivent les données. Elles répondent à des questions simples mais cruciales : Qu’est-ce que c’est ? Comment cela a été produit ? Dans quelles conditions ? Par qui ? Pourquoi ?
Un standard universellement reconnu a été établi pour toutes les données relevant de l'imagerie, le standard REMBI. Ce standard indique quelles sont les métadonnées nécessaires à la réutilisation des données d'imagerie dans d'autres disciplines ou à des fins de machine-learning.
Un fichier Excel complet téléchargeable regroupant toutes les métadonnées théoriquement nécessaires (il est d'ailleurs mentionné dans l'onglet "Notes" qu'il ne s'agit pas d'une implémentation directement utilisable) se trouve ici
Les métadonnées qui seront demandées sont très fortement inspirées de ce standard, mais dans un souci plus pratique, nous nous réfèrerons au site de l' EBI, plus centré sur la microscopie.